DIAG4ZOO se positionne en tant qu’expert dans le domaine de l’analyse du microbiome issu d’échantillons provenant de productions aquacoles.
Dans un contexte de changement climatique, de sécurité alimentaire et de suivi des populations, l’analyse du microbiome en aquaculture joue un rôle majeur dans la prévention des maladies ou des mortalités. De plus, l’impact du microbiome s’étend bien au-delà, car il joue un rôle déterminant dans la qualité de vie des poissons. En effet, la santé du microbiome influence directement la croissance des poissons ainsi que la qualité de leur chair, des aspects cruciaux dans le secteur de la production aquacole.
DIAG4ZOO a ainsi été missionnée par un grand producteur aquacole européen pour étudier l’écosystème de ses bassins, c’est-à-dire analyser l’ensemble des micro-organismes (bactéries, virus, champignons, levures) prédominants dans le milieu étudié.
Pour réaliser cette analyse du microbiome, DIAG4ZOO a suivi une approche intégrative des communautés virales et bactériennes (microbiomes) présentes dans différents échantillons tels que les œufs, les larves et les poissons (branchies et nageoires par prélèvements non létaux). De plus, l’environnement aquatique de l’animal, à savoir l’eau, fait également l’objet d’une analyse méticuleuse.
L’analyse du microbiome permet d’identifier et de dénombrer les micro-organismes en isolant les génomes présents dans les échantillons prélevés (eau, larve, fèces…) afin de les séquencer et d’identifier à quels organismes ces génomes sont associés.
Le processus d’analyse métagénomique suivi par DIAG4ZOO se déroule selon plusieurs étapes méthodiques, assurant une précision et une exhaustivité maximales. La méthode suivie est constituée des différentes phases successives suivantes :
Méthode d’analyse du microbiome
Ces analyses de métagénomique sont réalisées en plusieurs étapes :
- Extraction et purification des ADNs : cette étape vise à isoler de manière efficace le matériel génétique nécessaire à l’analyse.
- Qualification des ADNs : l’évaluation de la qualité des ADNs permet d’éliminer tout élément indésirable et de garantir la fiabilité et la reproductibilité des données génétiques à analyser.
- Amplification par PCR et séquençage des ADNs : les ADNs qualifiés sont amplifiés par réaction de polymérisation en chaîne (PCR), puis soumis à un séquençage à haut débit sur une plate-forme de dernière génération. Cette étape permet d’obtenir une quantité considérable de données génétiques à analyser.,
- Analyses bio-informatique et biostatistique des données de séquences : les données de séquences obtenues sont soumises à une analyse rigoureuse, combinant des techniques bio-informatiques avancées et des méthodes biostatistiques propriétaires. Cela permet de déchiffrer les informations génétiques et d’extraire des conclusions significatives..
En fonction des objectifs attendus et de la finalité de ces derniers (contrôle inopiné ou mise en place d’un outil de suivi en routine), DIAG4ZOO adopte une approche ciblée. Son équipe technique peut choisir de se concentrer sur une région variable du gène de l’ARN ribosomal 16S ou d’analyser l’intégralité de l’ADN ribosomique.
Dans la présente étude, la première approche (séquençage du gène de l’ARN ribosomal 16S) a été privilégiée. Les séquences métagénomiques ciblées provenant du microbiome ont été analysées en utilisant le pipeline bioinformatique élaboré par DIAG4ZOO.
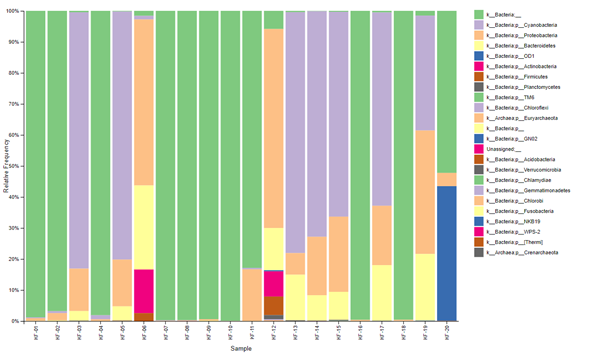
Ce travail a abouti à l’identification de la structuration des communautés procaryotes, également connues sous le terme d’unités taxonomiques opérationnelles (OTU), au sein des divers échantillons.
Le résultat final offre une classification des lectures à plusieurs niveaux taxonomiques, englobant le phylum, la classe, l’ordre, la famille, le genre et l’espèce, fournissant ainsi une compréhension approfondie de la diversité microbiologique présente dans les échantillons étudiés.
Ainsi, DIAG4ZOO, à travers son expertise approfondie et son approche intégrative de l’analyse du microbiome, contribue de manière significative à l’amélioration de la santé et de la productivité des poissons d’élevage, répondant ainsi aux défis contemporains liés à la durabilité, à la qualité alimentaire et à la gestion raisonnée des ressources aquatiques.
Plus généralement, l’équipe scientifique de DIAG4ZOO possède une expertise et une expérience importante dans les approches de Barcoding et de Metabarcoding, deux techniques sophistiquées basées sur l’extraction et le séquençage à haut débit (séquençage NGS) de l’ADN.
A ce sujet, l’équipe DIAG4ZOO excelle dans l’art de l’extraction minutieuse de l’ADN contenu dans divers échantillons, qu’ils soient homogènes ou hétérogènes. En utilisant ensuite des technologies de séquençage à haut débit, l’équipe parvient à obtenir une quantité significative de données génétiques de manière rapide et précise.
Ces approches de Barcoding et de Metabarcoding se révèlent particulièrement puissantes pour l’analyse d’une variété d’échantillons, même hétérogènes, ouvrant ainsi la voie à de multiples applications :
- Détection rapide et spécifique d’agents pathogènes présents dans des échantillons, permettant de renforcer les actions de prévention et de gestion des maladies ;
- Surveillance des Espèces Invasives, offrant des informations cruciales pour la préservation des écosystèmes ;
- Suivi détaillé des Groupes Taxonomiques, permettant ainsi une meilleure compréhension de la diversité biologique au sein des échantillons ;
- Etude des Régimes Alimentaires, fournissant des informations précieuses sur les interactions écologiques et les chaînes alimentaires.
L’expertise de DIAG4ZOO dans les techniques de Barcoding et de Metabarcoding peut ainsi contribuer significativement à l’avancement des connaissances dans des domaines cruciaux tels que la santé environnementale, la préservation de la biodiversité et la sécurité alimentaire.
Pour en savoir plus sur l’expertise de DIAG4ZOO dans l’analyse du microbiome, les analyses de métagénomique, les approches de Barcoding et de Metabarcoding, dans le séquençage des génomes ou ADNs, en aquaculture et dans des échantillons complexes, contactez notre équipe commerciale.
